Table of Contents
Cvičení 3 : Registrace
Na třetím cvičení se budeme zabývat registrací obrazů. Ta je potřebná ve chvíli, kdy máme dva obrázky <latex>I, J</latex> (např. rozdílné modality, rozdílné časy vytvoření snímku, apod.) a chceme informace z těchto dvou vstupů spojit dohromady. Cílem je tedy nalézt transformaci <latex>T</latex>, která převede obrázek <latex>J</latex> na obrázek I s co nejlepším možným překryvem.
Na cvičení budeme pracovat s matlabem a vyzkoušíme si jak manuální, tak i automatickou registraci různých obrázků.
Zadání
- Landmark registration Pracujte s dvojicí obrázků
histology.HEstainahistology.PanCytokeratin, které zobrazují skoro identický řez ve dvou různých barveních (Hematoxylin+Eosin a Cytokeratin). Určete dostatečný počet kontrolních bodů a nejděte co možno nejlepší transformaci. Vyzkoušejte alespoň dvě transformace, ukažte výsledek a diskutujte volbu transformace, rozmístění kontrolních bodů ve zprávě [1b]
- Automatic registration Máme k dispozici dva MRI snímky (stejného pacienta) -
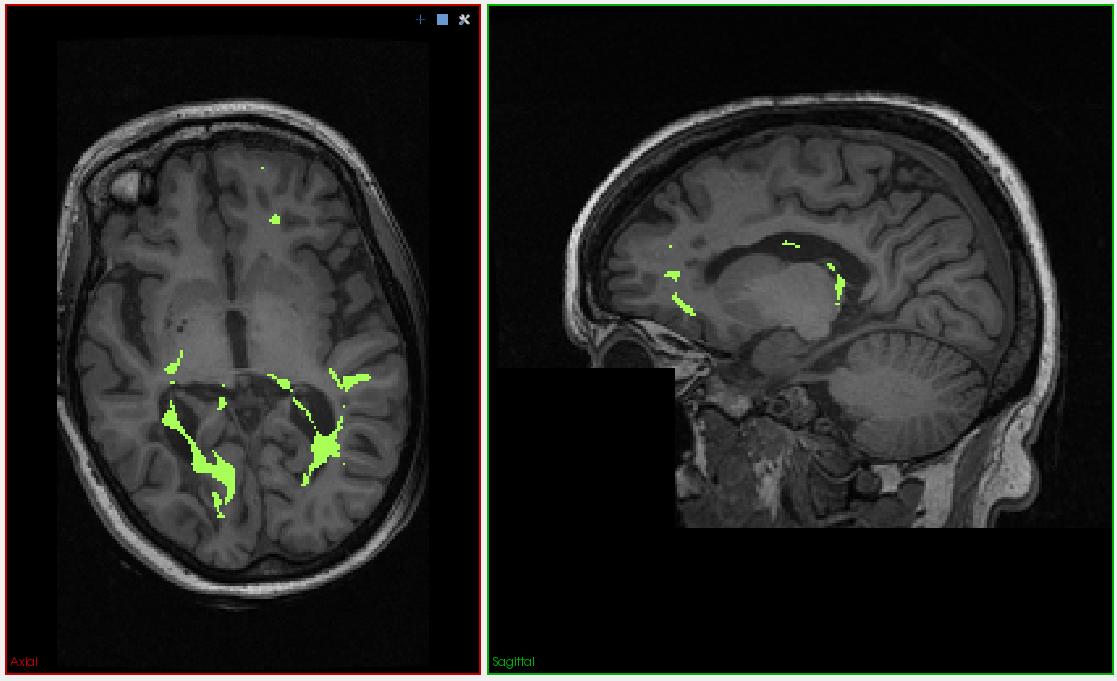
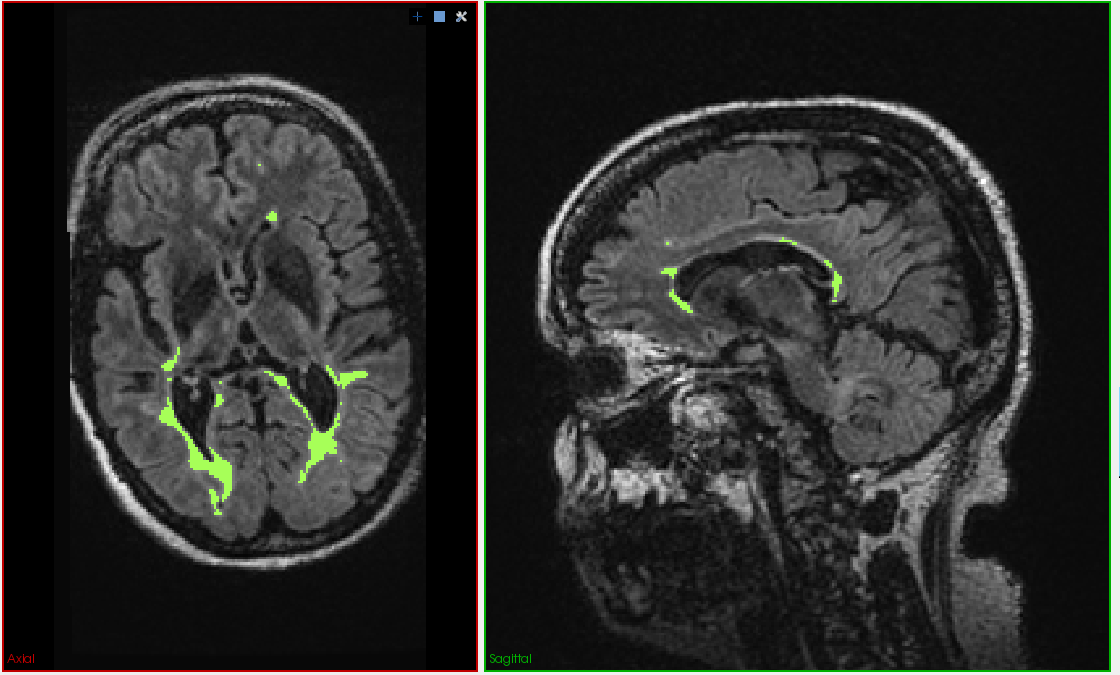
T1.niiaFLAIR.nii, a dále segmentaci lézí na snímku FLAIR - obrázekLesionSeg.nii. Spočítejte použitím automatické registrace transformaci snímku FLAIR na snímek T1 a touto pak přetransformujte snímek segmentací. [2b] Spočtěte histogram intenzit snímku T1 přes segmentované oblasti. [BONUS +1b]
- Deformable registration Zde máme dva MRI snímky kolene od různých pacientů -
58_MRI.niia64_MRI.nii. Spočítejte použitím automatické registrace transformaci snímku 64 na snímek 58. Protože se jedná o dvě rozdílné anatomie, musíme použít metodu pružné a konzistentní deformace, abychom dosáhli rozumného zarovnání. [2b]
Data, funkce
Vstupní data a pomocné funkce pro načítání a zobrazování jsou ke stažení jako ZIP Archiv. V archivu je adresář registration_images a cv3_matlabfun. Po rozbalení je potřeba přidat adresář cv3_matlabfun v Matlabu (add path), pak jsou funkce dostupné.
- Načítání obrázků
fixed = imread('registration_images/brain.t1.png');
- Načítání 3D obrázků funguje za pomocí metody
load_niiimage1 = load_nii('58_MRI.nii'); fixed = image1.img;
Funkce vytvoří struct image1, podimage1.imgse pak nachází obrazová data. - Zobrazení dvou obrázků imshowpair
figure, imshowpair( m_poly, fixed );
pro 3D obrázky si lze buď zobrazit n-tou vrstvu pomocíimage(:,:,n)nebo použijte metoduimshow3Dz přiloženého balíčku pomocných funkcí.imshow3D(moving, fixed, 'montage', 1);
Parametry funkceimshow3Djsou dva obrázky, které chceme zobrazit, dále metoda překryvumontage|falsecolor|blenda jako poslední dimenze, kterou chceme zobrazit.
Landmark registration
Registrace pomocí kontrolních bodů je jednou ze základních poloautomatických metod. Začneme tím, že ručně určíme páry bodů <latex> (x_i, y_i), i=1, \ldots N </latex> pro významné pozice, které lze identifikovat v obou obrázcích a pak hledáme transformaci <latex>T</latex>, která minimalizuje vzdálenost <latex> x_i </latex> a <latex> T(y_i) </latex>.
Postup
- Spustíme interaktivní rozhraní cpselect pro definici kontrolních bodů
h = cpselect(moving, fixed);
- Před zavřením dialogového okna je potřeba uložit páry bodů do souborů, např
fixedPointsamovingPoints - S těmito množinami bodů pak pracujeme dále a hledáme optimální transformaci pomocí fitgeotrans
t_sim = fitgeotrans(movingPoints, fixedPoints, 'similarity');
Typ transformace (v příkladusimilarity) je závislý na vstupních datech, možné další třídy transformací jsou v uvedené v dokumentaci - Nakonec transformujeme
movingimage spočtenou transformacím_sim = imwarp(moving, t_sim, 'OutputView', imref2d(size(fixed)));
Poslední argumentimref2dspecifikuje, že se vstupní obrázek má transformovat na mřížku o velikosti vstupního obrázkufixed.
Automatic registration
Příklad s MRI snímky hlavy je typickým problémem při vyhodnocování medicínským obrazů. K jednomu pacientovi máme dva různé snímky – MRI sekvence T1 a FLAIR. Na druhém jsou dobře vidět léze jako světlé oblasti a zajímají nás hodnoty druhém obrázku ve stejných oblastech. Kvůli pohnutí hlavy mezi snímáním jednotlivých sekvencí nejsou obrázky zarovnány a tudíž momentálně nesouhlasí segmentace a T1 snímek, jak je zřejmé z levého obrázku následující ilustrace.
Metoda
Automatická registrace v MATLABu je dostupná přes funkci imregister (vrací přetransformovaný obrázek) nebo imregtform (vrací transformaci). Abychom ji mohli spočíst, musíme specifikovat postup optimalizace přes parametr optimizer a dále pak vhodnou metriku, která měří chybu zarovnání – parameter metric. Jedná se o snímky s rozdílnými kontrasty, proto použijeme metriku MutualInformation
registration.metric.MattesMutualInformation Properties: NumberOfSpatialSamples: 500 NumberOfHistogramBins: 50 UseAllPixels: 1Nastavení parametrů uvedených pod
Properties speficikují, jakým způsobem se metrika vyhodnocuje a má výrazný vliv na průběh a výsledek registrace. Výpočet lze urychlit, pokud nebudeme používat všechny pixely (UseAllPixels = 0), pak ale musíme počítat metriku přes dostatečné množství vzorků (NumberOfSpatialSamples = 50000 a více). Posledním parametrem před spuštěním registrace je nastavení třídy transformací. Jedná se o stejného pacienta, tudíž bychom mohli použít rigidní transformaci, lepší volbou je affiní transformace, která spolu s pohybem hlavy do určité míry kompenzuje rozdílná zkreslení v sekvencí i T1 a FLAIR.
Objekt optimizer získáme následovně
opt_gd = registration.optimizer.RegularStepGradientDescent opt_oneplus = registration.optimizer.OnePlusOneEvolutionary
Postup
- Načteme soubory
brain_T1.niiabrain_FLAIR.niipomocí funkceload_niiobsažené v balíčku s pomocnými funkcemi, od verze R2017b lze použít příkazniftyread - Nastavíme objekty
metricaoptimizera zavoláme funkci pro registraci obrázkůmoving_transformed = imregister(moving, fixed, 'rigid', optimizer, metric); % pokud počítáme transformaci, pak použijeme moving_t = imregtform(moving, fixed, 'rigid', optimizer, metric); moving_transformed = imwarp(moving, moving_t, imref3d(size(fixed)));
- Výslednou transformaci aplikujeme na FLAIR image, abychom zjistili kvalitu registrace, pokud je dobrá, pak můžeme transformaci aplikovat i na snímek se segmentací
- Histogram spočteme pomocí funkce imhist, nejprve ale musíme aplikovat transformovatnou masku
Deformable registration
Při registraci obrázků s rozdílnou anatomií není affiní transformace dostačující a je potřeba registrovaný obrázek deformovat. Tato metoda se používá ve chvíli, kdy chceme z množiny snímků vytvořit atlas (tj. mít obrázek, jak v průměru daná anatomická struktura vypadá), nebo když nás zajímá, jak se tkáň mezi dvěma snímky získaných s delším časovým odstupem změnila.
Abychom nemuseli počítat příliš velké deformace, spočteme v prvním kroku affiní transformaci (viz. předchozí úloha) a deformaci budeme určovat až mezi výsledkem affiní registrace a cílovým obrázkem
Postup
- Spočteme
moving_affpomocí funkceimregister. Snímky mají stejný rozsah intenzit, použijeme tedy metrikuregistration.metric.MeanSquare, optimalizovat budeme pomocíregistration.optimizer.RegularStepGradientDescent - Spočteme deformaci mezi
moving_affafixedpomocí metodyimregdemons[D, moving_def] = imregdemons( moving_aff, fixed, [400 150 60], 'AccumulatedFieldSmoothing', 1.8);
Program pracuje s tzv. pyramidou - nejprve počítá deformaci na vstupech s redukovabným rozlišením, takto spočtenou deformaci postupně zjemňuje na úrovních pyramidy se rostoucím rozlišením, poslední úroveň je pak vstupní obrázek. Parameter[400 150 60]určuje, kolik iterací se počítá na jednotlivých úrovních pyramidy. Druhý parameter -AccumulatedFieldSmoothingregularizuje výslednou deformaci. Menší hodnoty umožňí detailnější deformace, vyšší hodnoty zvyšují hladkost výsledného deformačního pole.
Vizualizaci výsledné deformace můžeme získat aplikací deformačního pole na obrázek se 3D-šachovnicí s velikostí odpovídající vstupním obrázkům.
slice = checkerboard( 32, 4, 4 ); for i = 1:1:60 if mod( floor(i / 10), 2) > 0 check(i, :, :) = uint8(255 * slice(:, :)); else check(i, :, :) = uint8( 255 * (1 - slice(:, :))); end end warped_checkerboard = imwarp( check, D ); % Zobrazíme deformovaný vstup a vizualizaci pole vedle sebe imshow3d( moving_def, warped_checkerboard, 'montage', 1);